Vol. 2° - III.6.2.
Trascrizione del DNA
Il genoma di un organismo è
costituito da sequenze di coppie di basi distribuite sui cromosomi. Tutte le
coppie di basi del genoma vengono replicate durante la fase di sintesi del
DNA, mentre soltanto alcune coppie di basi vengono trascritte in RNA.
Le sequenze di coppie di basi che vengono trascritte prendono
il nome di geni, per cui il processo di trascrizione viene anche detto
espressione
genica.
All’inizio e della fine di ogni gene esistono sequenze dette elementi genici regolatori,
coinvolte nella regolazione dell’espressione genica.
La trascrizione genera quattro
classi principali trascritti, o di molecole di RNA:
§
RNA messaggero «
mRNA
§
RNA transfer «
tRNA
§
RNA ribosomiale «
rRNA
§
il piccolo RNA nucleare, small nuclear RNA «
snRNA.
Procarioti ed eucarioti posseggono entrambi l’RNA
messaggero, il tRNA e l’rRNA, mentre l’snRNA si trova solo negli
eucarioti. Per il momento è importante ricordare che soltanto le molecole di mRNA vengono tradotte in proteine.
Un gene che codifica per una molecola di mRNA, e quindi
per una proteina, viene detto gene
strutturale. Esistono trascritti di altri
geni sotto forma di RNA che rappresentano prodotti finali dell’espressione
genica e che non fungono come molecole messaggere tra DNA e proteina, cioè,
non vengono mai tradotti in proteine. I geni che codificano per il tRNA, l’rRNA
e l’snRNA appartengono a questo tipo di geni.
I geni sono quasi sempre localizzati sui cromosomi, e
rimanendo nel nucleo devono essere in grado di controllare le funzioni in
tutta la cellula. Essi, attraverso la trascrizione, producono dei trascritti
che possono abbandonare i cromosomi ed essere coinvolti nella sintesi proteica
in altre parti della cellula.
Il DNA è una struttura stabile che si ritrova pressoché
immutata al termine della riproduzione cellulare, mentre i trascritti di mRNA
dovuti ai geni hanno un’esistenza limitata nel
tempo, essendo degradati da enzimi cellulari, le ribonucleasi.
Il mantenimento temporaneo dei trascritti di RNA è un elemento chiave nel
processo di regolazione dell’espressione genica.
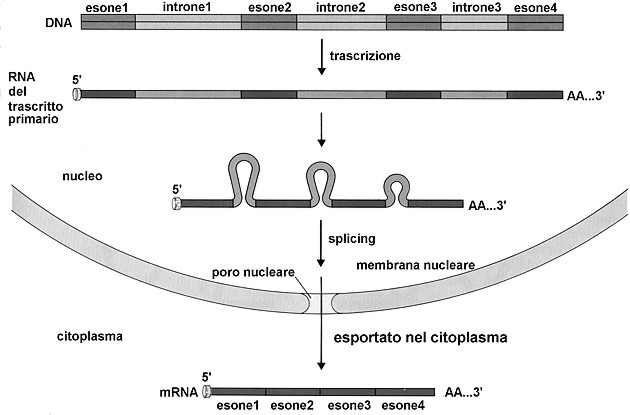
Fig. III. 27 - Splicing di un trascritto primario. L’RNA messaggero viene prodotto nel nucleo cellulare, sul modello di un filamento di DNA, per mezzo di un enzima denominato RNA polimerasi DNA dipendente. Sempre nel nucleo, l’mRNA primitivo così costituitosi viene sottoposto ad un rimaneggiamento, detto splicing, che consiste essenzialmente nell’eliminazione degli introni, privi di funzione codificante. L’mRNA maturo viene trasferito nel citoplasma e sistemato sui ribosomi dove viene letto.
6.2.a. Caratteristiche fondamentali della trascrizione
Nei procarioti e negli eucarioti
la trascrizione avviene grazie a un processo biochimico catalizzato dalla RNA polimerasi.
Prima che la trascrizione possa cominciare, la doppia elica di DNA deve
essersi srotolata. Soltanto
una delle due eliche di DNA viene trascritta in RNA. Questa elica è
detta elica stampo,
e l’elica che non funge da stampo, cioè quella complementare, ha la stessa
polarità della catena di RNA.
Se tutt’e due le eliche di DNA di un gene venissero
trascritte in molecole di RNA, ogni gene produrrebbe due RNA con sequenze tra
loro complementari e la traduzione dei due RNA darebbe origine a due proteine
molto diverse. Prove di genetica indicano invece che ogni gene strutturale
codifica per una sola proteina e che accade quanto previsto, cioè che da
parte di un dato gene viene trascritta una sola molecola di RNA.
Nel processo di trascrizione i precursori dell’RNA sono i ribonucleosidi
trifosfati ATP,
GTP, CTP e UTP,
indicati come NTP
(nucleosidi trifosfati L’enzima che catalizza la reazione di
polimerizzazione è la RNA polimerasi, specifico per la sintesi dell’RNA,
che utilizza soltanto NTP contenenti ribosio, e non quelli contenenti
desossiribosio che è presente nei precursori del DNA. La polimerizzazione
dell’RNA è molto simile a quella del DNA. Il nucleotide successivo da
inserire nella catena è selezionato dalla RNA polimerasi per la sua capacità
di appaiarsi alla base esposta sull’elica stampo di DNA. Si ricordi che le
catene di RNA contengono nucleotidi con la base uracile anziché timina.
Pertanto, quando si presenta sull’elica stampo del DNA un nucleotide A,
verrà inserito sulla catena di RNA un nucleotide U.
6.2.b. Trascrizione del DNA
La trascrizione del DNA è
attiva durante tutti i momenti dell’interfase e corrisponde alla sintesi di una molecola di RNA a
partire da un solo filamento di DNA che non sia eterocromatina, in quanto il
DNA dell’eterocromatina non viene trascritto perché non contiene sequenze
di geni (eterocromatina costitutiva), o perché contiene sequenze di geni
inattivi (eterocromatina facoltativa). Solo
il DNA dell’eucromatina viene trascritto.
La trascrizione consiste nel processo di lettura delle sequenze a doppia elica del DNA
in sequenze
a singola elica di RNA: mRNA, tRNA, e rRNA in procarioti ed eucarioti, e
nei soli eucarioti si verifica anche la sintesi di snRNA.
La trascrizione è un processo che presenta
caratteristiche simili in procarioti ed eucarioti: il DNA si denatura e la RNA
polimerasi catalizza la sintesi di una molecola di RNA in direzione 5’ ® 3’. Soltanto una delle due
eliche di DNA viene trascritta nella molecola di RNA a singola elica. Sequenze promotrici
e terminatrici stabiliscono
rispettivamente dove debba cominciare e finire la trascrizione.
In Escherichia coli
la sola RNA polimerasi trascrive tutte le diverse classi di RNA della cellula
e cioè l’mRNA, il tRNA e l’rRNA. Nei nuclei delle cellule eucariotiche
esistono tre RNA
polimerasi con funzioni diverse: la RNA polimerasi I
trascrive tre dei quattro rRNA, la RNA
polimerasi II
trascrive gli mRNA e alcuni snRNA mentre la RNA
polimerasi III
trascrive il quarto rRNA, i tRNA e i rimanenti snRNA.
A differenza della RNA polimerasi di Escherichia coli, nessuna delle tre polimerasi eucariotiche si lega
direttamente al DNA durante la fase iniziale della sintesi dell’RNA. L’inizio
della trascrizione da parte di questi enzimi è mediata da fattori
trascrizionali che sono specifici per ciascuna polimerasi e che riconoscono sequenze promotrici appropriate.
Il
promotore per i geni che codificano proteine è localizzato a monte
rispetto al punto d’inizio della trascrizione ed è costituito da
combinazioni diverse di elementi promotori. Fattori trascrizionali specifici e
fattori regolatori si legano a questi elementi e facilitano l’inizio della
trascrizione da parte della RNA
polimerasi II. In posizione più distale rispetto al promotore si trovano
delle sequenze enhancer
(intensificatori), alle quali si legano dei fattori regolatori che hanno la
funzione di facilitare al massimo la trascrizione genica.
I promotori dei geni trascritti dalla RNA polimerasi III
sono localizzati all’interno della sequenza genica codificante e consistono
in combinazioni diverse di dominî funzionali, caratteristici per la classe di
RNA che viene trascritta. Questi dominî vengono riconosciuti dai fattori
trascrizionali, che facilitano l’inizio della trascrizione della RNA
polimerasi.
Gli rRNA 18S, 5.8S e 28S vengono trascritti in un’unica
unità trascrizionale, producendo una molecola di RNA precursore. La maggior parte degli eucarioti ha le unità
trascrizionali ripetute in serie, una di seguito all’altra, ripetute
in tandem, separate da una sequenza
spaziatrice non trascritta (nontranscribed spacer
o NTS). Il promotore per l’unità
trascrizionale è localizzato nell’NTS al quale si legano fattori
trascrizionali specifici, che facilitano l’inizio della trascrizione dell’RNA polimerasi I.
Il
materiale genetico possiede tre caratteristiche fondamentali:
ü
Contiene,
in forma stabile, tutte le informazioni necessarie per determinare la
struttura dell’organismo, le sue funzioni, lo sviluppo e la riproduzione.
ü
Si
replica in modo accurato, affinché le cellule figlie possano avere la stessa
informazione genetica della cellula madre.
ü
È
capace di variare.
Passiamo ad esaminare i processi mediante i quali il
materiale genetico dirige la sintesi proteica. Poiché tutte le funzioni
cellulari comportano l’azione di proteine specifiche, in ultima analisi tali
funzioni vengono governate dal materiale genetico. Ogni proteina è formata da
una o più catene di aminoacidi, codificati da una sequenza specifica di
coppie di basi del DNA. Quando la cellula ha bisogno di una certa proteina, l’informazione
genetica per la sequenza aminoacidica di tale proteina dev’essere ricavata
dal DNA e quindi trasformata nel prodotto finito.
La sintesi proteica comporta due tappe:
·
trascrizione: è il processo di
trasferimento dell’informazione dalla molecola stampo a doppia elica di DNA
a una molecola di RNA a elica singola.
·
traduzione o sintesi proteica: è la trasformazione in una sequenza di
aminoacidi dell’informazione codificata in seno alla sequenza di basi dell’RNA
messaggero.
Mentre
la duplicazione del DNA ha luogo soltanto in una fase del ciclo cellulare,
negli eucarioti la trascrizione e la traduzione dei geni si verifica durante tutto il ciclo
cellulare.
6.2.c. Meccanismo della trascrizione
La RNA polimerasi DNA dipendente è l’enzima che
assicura la formazione di una molecola di RNA attraverso il concatenamento di
nucleotidi in un ordine che è predeterminato dalla molecola di DNA. Così,
per una molecola di DNA caratterizzata dalla sequenza:
CGG
ACT TTC
GTG
la sequenza nucleotidica dell’RNA
sarà ad essa complementare:
GCC UGA AAG
CAC
I nucleosomi non esercitano alcun ostacolo sulla
trascrizione del DNA. Le RNA polimerasi riconoscono la fibra di DNA a tutti i
livelli del filamento che si trova avvolto ad elica intorno ai nucleosomi. Il
filamento nucleosomico soggiace ad alcune modificazioni: gli ottameri di
istoni si suddividono in tetrameri, generando un filamento detto eminucleosoma e ogni eminucleosoma è circondato da un avvolgimento
di DNA. La trascrizione è preceduta da un’acetilazione degli istoni e le
proteine acide intervengono nella struttura funzionale del nucleofilamento per
permettere l’espressione dei geni che sono programmati per ciascun tipo
cellulare.
6.2.d. Tappe della trascrizione
Riconoscimento del promotore
Il promotore è una speciale
sequenza di nucleotidi situata sul DNA all’inizio del gene. L’RNA
polimerasi si fissa sul promotore grazie al polipeptide s (sigma). In sua assenza sembra che vi sia comunque l’inizio
della lettura, ma in un punto qualsiasi del gene.
Inizio della sintesi
La sintesi di RNA inizia in un
punto fisicamente diverso dal promotore: si tratta del sito d’inizio. Non appena la RNA polimerasi
si lega a questo sito, il polipeptide s
si allontana, e la lettura e la sintesi vengono assicurate dal nucleo centrale
della polimerasi.
Copiatura del gene
La RNA polimerasi legge
un filamento della molecola di DNA spostandosi nel senso 3’ ®
5’. Per giungere alla trascrizione del DNA in RNA è necessaria la selezione
di precursori, che consistono in nucleosidi complementari appropriati, nonché
la formazione di legami fosfodiesterici fra nucleotidi adiacenti.
I precursori, sotto forma di nucleosidi 5’ trifosfati, sono i
seguenti:
q
adenosina
5’ trifosfato: ATP
q
citidina
5’ trifosfato: CTP
q
guanosina
5’ trifosfato: GTP
q
uridina
5’ trifosfato: UTP
Crescita della molecola di RNA
Via via che la molecola di RNA
polimerasi si sposta lungo il filamento, i nucleotidi si uniscono fra loro con
un legame fosforico 3’ Ö
OH ¾ 5’ Ö OH.
Fine della sintesi
La sintesi si arresta a livello
di determinati siti della molecola del DNA, che costituiscono i segnali
di termine della lettura. L’arresto della sintesi avviene solo in presenza
di una proteina di peso molecolare 50.000, il fattore r (rhota). Alla fine della lettura la molecola di RNA e la
RNA polimerasi DNA dipendente si allontanano dal DNA. L’mRNA neosintetizzato
compare nel nucleo sotto forma di fibrille pericromatiche.
La trascrizione del DNA non
porta solo alla formazione di mRNA
che è una copia del gene,
ma anche alla sintesi di rRNA e di tRNA che sono indispensabili alla
traduzione dell’mRNA, ma che non vengono tradotti in proteine.
Fatta eccezione per l’mRNA, i prodotti della
trascrizione, prima di essere utilizzati, vanno incontro a modificazioni post-trascrizionali.
Per esempio, l’rRNA, che si forma unicamente nel nucleolo, è una lunga
molecola: rRNA 45S; subito dopo essere stato trascritto, viene metilato ad
opera di metilasi e viene scisso da endonucleasi, passando alle forme 16S, 23S
e 5S.
6.2.e. Regolazione della trascrizione
Le cellule regolano la sintesi
di RNA in base alle loro esigenze e alle variazioni del mezzo ambiente. Per
tale adattamento è necessario un controllo dell’attività dei geni, che
avviene sia modificando la capacità dell’RNA polimerasi di riconoscere il
sito promotore, sia agendo su un sito vicino al promotore, detto sito
operatore. Tali meccanismi di regolazione sono ben conosciuti nei batteri.