Vol. 2° - III.7.1.
Meiosi
prima
La prima divisione meiotica
Le cellule germinali maschili, gli spermatogoni, o femminili, gli oogoni, si sono riprodotte un certo numero di volte per mitosi, restando quindi diploidi.
Alcune di esse si preparano alla meiosi e in tal caso entrano in una
fase S più lunga del solito, che si prolunga fino all’inizio della profase.
Queste cellule prendono ora il nome di spermatociti
e oociti.
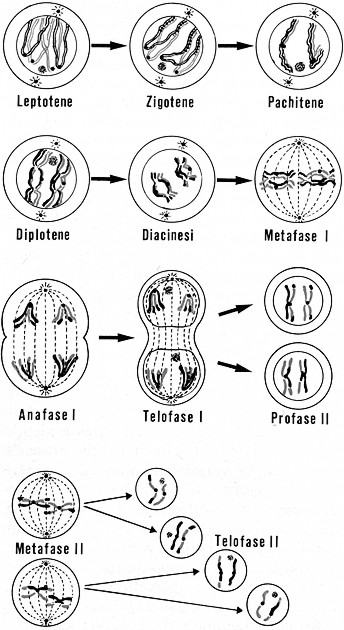
Fig.
III. 30
- Le fasi della meiosi in una cellula con dotazione diploide pari a 4
cromosomi.
Al
termine del processo meiotico, da una cellula diploide otterremo 4 gameti
aploidi.
7.1.a. Profase 1ª
Nella profase prima i cromosomi si accorciano e si ispessiscono, si verifica il crossingover, si forma l’apparato del fuso, membrana nucleare e nucleolo - o nucleoli - si dissolvono. La profase 1ª è molto lunga, tanto da meritare il nome di profase meiotica per eccellenza essendo la profase 2ª brevissima, ed è suddivisa in alcuni sottostadi. Prende inizio dopo la replicazione dei cromosomi.
Inizio della profase « stadio di leptotene.
In questo stadio i cromosomi
hanno cominciato a spiralizzarsi e diventano visibili come strutture
filamentose, più lunghi di quelli della profase mitotica. Non sono altro che
i cromatidi fratelli tenuti insieme dal centromero. Il numero di cromosomi in
questo stadio corrisponde a quello di una cellula diploide. Quando la cellula
entra nello stadio di leptotene si impegna in modo definitivo nella meiosi.
Inizio della fase media « stadio di zigotene.
Nello zigonema i cromosomi
omologhi cominciano ad appaiarsi in modo altamente specifico, punto per punto,
come i due elementi di una chiusura lampo.
Questo appaiamento è detto sinapsi. Poiché la replicazione è già avvenuta,
ciascuna serie di cromosomi sinaptici è costituita da 4 cromatidi e viene
indicata col termine di serie bivalente
o tetrade.
Fase media della profase « stadio di pachitene.
Allo zigonema segue il
pachinema, fase in cui i cromosomi sono molto più corti e spessi, e sono
visibili come strutture a quattro filamenti intimamente appaiati. Durante il
pachinema avviene l’evento più importante, il crossingover, cioè lo scambio reciproco tra cromosomi omologhi
di segmenti cromosomici localizzati nella stessa posizione lungo il cromosoma.
Se esistono differenze genetiche tra gli omologhi, il crossingover produce in un cromatide nuove combinazioni geniche. Il crossingover non causa né perdita né acquisizione di materiale genico, poiché determina solo scambi reciproci.
Lo scambio consta in una frattura, in punti corrispondenti di cromatidi non
fratelli, di una porzione di materiale genetico, nel suo scambio con l’equivalente
porzione dell’altro cromatidio e quindi nella risaldatura nella nuova
posizione del pezzo scambiato. I meccanismi di scambio sono poco evidenziabili
perché, mentre si svolgono, i quattro cromatidi della tetrade pachitenica
sono strettamente affiancati.
Il crossingover è facilitato
dall’unione sinaptica stretta, cioè un allineamento fianco a fianco dei 4
cromatidi, che porta alla formazione di strutture simili a cerniere lampo per
tutta la loro lunghezza.
L’appaiamento punto per punto di ciascuna coppia di omologhi
risulta diretto da una particolare struttura proteica che compare in zigotene,
perdura in pachitene e scompare più tardi: si tratta del complesso sinaptinemale,
formazione nastriforme interposta fra gli omologhi, che quindi non sono a
diretto contatto, composta da tre zone disposte in parallelo lungo l’asse
longitudinale del complesso.

Fig.
III. 31 -
Evoluzione del complesso sinaptinemale durante la profase della meiosi.
C1+2 sono i cromatidi fratelli 1 e 2 - C3+4 sono gli altri cromatidi fratelli 3 e 4
1 formazione centrale interposta fra gli omologhi - 2 nodulo di
ricombinazione.
Tale struttura nastriforme è soprattutto costituita da
proteine acide alle quali è adeso dell’RNA, ma non si conosce il ruolo che
esso svolge nel crossingover. Tali proteine si dispongono a formare unità
globulari, dette noduli di ricombinazione,
presenti nell’elemento centrale del complesso sinaptinemale. In alcuni
organismi, come nei maschi di Drosofila, il complesso sinaptinemico non si
forma e in questi casi il crossingover non avviene.
Probabilmente, enzimi del tipo delle endonucleasi spezzano il DNA dei cromatidi non fratelli in punti corrispondenti e infine enzimi del tipo delle ligasi saldano i tratti scambiati. Il cromosoma che esce dalla meiosi con una combinazione di geni differente rispetto alla combinazione di partenza viene detto cromosoma ricombinante.
Il crossingover è quindi un meccanismo in grado
di produrre ricombinazione genica, un concetto che esamineremo in dettaglio
successivamente. Comunque, dopo il crossingover, almeno uno dei due cromatidi
di ciascun omologo formante un bivalente risulta geneticamente differente
rispetto alla situazione iniziale.
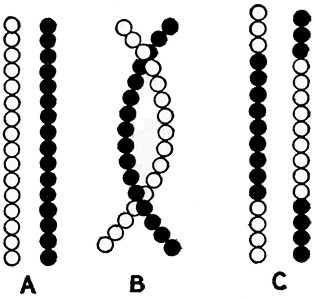
Fig.
III. 32 -
Interscambio o crossingover
Schema molto semplificato del prodotto finale
dovuto al crossingover: due cromosomi ricombinanti
Fase medio-terminale della profase « stadio di diplotene
Nella fase di diplonema i cromosomi cominciano a separarsi, però nei punti dove non c’è stato crossingover, in quanto in questi punti rimangono in contatto tra loro. Il processo di crossingover diventa visibile con la formazione di strutture cruciformi, a occhielli, denominate chiasmi, in quanto somigliano alla lettera greca chi c, e i punti di contatto corrispondono a quelli precedentemente interessati dallo scambio.
Poiché tutti e quattro i cromatidi
possono essere coinvolti in eventi di crossingover in qualsiasi punto degli
omologhi, la forma che il chiasma assume in questo stadio può essere molto
complicata, ed è molto usata dai citogenetisti perché fornisce indizi
precisi sull’entità degli scambi che si verificano nelle cellule indagate.
In molti organismi il diplonema è seguito rapidamente
dalle altre fasi della meiosi, ma gli
oociti di numerosi animali possono rimanere in fase di diplonema per parecchio
tempo. Per esempio, nella donna gli oociti giungono alla fase
di diplonema entro il settimo mese di vita endouterina e si arrestano in
questo stadio per parecchi anni. Dall’inizio della pubertà, e fino al
raggiungimento della menopausa, ad ogni ciclo mestruale matura un solo oocita
per ovocellula aploide, che viene quindi rilasciata dall’ovaio.
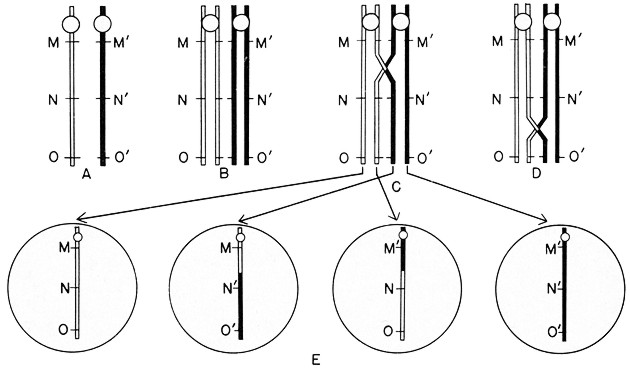
Fig. III. 33 - Interscambio in una coppia di cromosomi omologhi che sono eterozigoti per i loci M-N-O.
A
coppia di cromosomi omologhi - B stadio a 4 filamenti
C
interscambio tra due cromatidi non-fratelli nel tratto compreso tra i loci M e
N
D lo stesso interscambio nella regione compresa fra N e O
E
raffigura i 4 tipi di combinazioni cromosomiche possedute dai gameti e
derivate dal crossingover mostrato in C.
Fase terminale della profase « diacinesi
Durante questo stadio i 4
cromatidi di ciascun bivalente, o tetrade, è ancora più condensato e i
chiasmi si terminalizzano, cioè si
spostano verso le estremità dei cromatidi che così appaiono uniti in
posizione terminale. La terminalizzazione deriva forse dalla diversa
contrazione delle varie regioni cromosomiche e non da un reale spostamento dei
chiasmi. In questa fase si ha anche la scomparsa dei nucleoli e l’inizio del
dissolvimento della membrana nucleare. Si possono facilmente contare i
cromosomi e le tetradi si portano sul fuso per dare inizio alla metafase.
7.1.b. Metafase 1ª
All’inizio della metafase 1ª
la membrana nucleare si è completamente dissolta e le tetradi si allineano
sul piano equatoriale della cellula. L’apparato del fuso è ora
completamente formato e i suoi microtubuli sono attaccati ai centromeri degli
omologhi. È importante sottolineare che sulla piastra metafasica si trovano
le coppie di omologhi uniti sinapticamente, sono cioè presenti i bivalenti.
Invece, nella mitosi della maggior parte degli organismi,
i cromosomi omologhi replicati, cioè le coppie di cromatidi fratelli, si
allineano in modo indipendente sulla piastra metafasica. In altre parole, la
differenza fondamentale fra meiosi e mitosi è che nella meiosi i centromeri
fratelli rimangono uniti, mentre nella mitosi si separano.
7.1.c. Anafase 1ª
In questa fase i cromosomi di
ciascuna tetrade si separano in modo tale che le coppie di omologhi, i
bivalenti, separandosi possano migrare a poli opposti, dove si formeranno i
nuovi nuclei. In questo stadio ciascuna coppia è chiamata diade. L’orientamento verso l’uno o l’altro polo di ciascuna
diade è casuale. Per cui, anche prescindendo dal fatto che il crossingover ha
modificato l’assortimento genico di ogni cromosoma iniziale, ciascuna della
cellule figlie derivate dalla prima divisione meiotica riceve una serie di
cromosomi materni o paterni variamente combinati fra loro. Ciò contribuisce
ulteriormente a differenziare geneticamente i gameti fra loro.
Questa migrazione determina due fatti:
· gli omologhi di derivazione materna e paterna si separano
in modo casuale a ciascun polo, ad eccezione delle porzioni cromosomiche
scambiate durante il crossingover
· a ciascun polo si trova un complemento aploide per ciascun
cromosoma replicato.
È importante ricordare che in questo stadio le coppie di
cromatidi fratelli che hanno segregato rimangono attaccate tra loro attraverso
i rispettivi centromeri e che a ciascun polo della cellula in divisione si
sono portati assetti di diadi corrispondenti alla metà del numero diploide.
7.1.d. Telofase 1ª
La sua durata varia molto a
seconda delle specie. Ciò nonostante, nella telofase le diadi completano la
loro migrazione ai poli opposti della cellula e, nella maggior parte dei casi,
si formano nuove membrane nucleari attorno a ciascun raggruppamento aploide.
Nella maggiore parte delle specie si verifica la
citodieresi e vengono prodotte due cellule aploidi. La meiosi 1ª comincia
quindi con una cellula diploide, che contiene una serie cromosomica derivata
dalla madre e una derivata dal padre, e termina con due nuclei, ciascuno dei
quali contiene una serie di cromosomi replicati che possono essere sia di
origine paterna sia materna, ancora uniti al centromero. Dopo la citochinesi
ognuna delle due cellule figlie ha un nucleo con una serie aploide di
cromosomi replicati, quindi con una quantità 2c
di DNA, perché ogni cromosoma, o diade, è ancora costituito da due
cromatidi.
Dopo un’intercinesi breve o inesistente, ma comunque con la formazione di un nuovo apparato mitotico in ciascuna cellula figlia, inizia la profase della seconda divisione meiotica.